1.PUREfrexの合成量と鋳型DNAの配列について
PUREfrex を用いてタンパク質を合成した場合、その合成量が、合成するタンパク質の種類によって異なることはよく見られる現象です。この原因のひとつとして、そのタンパク質のアミノ酸配列に由来することが考えられ、これまでに、アミノ酸配列を最適化することで、合成量が改善されるという結果を得てきました。
アミノ酸最列の最適化は、おもに、下記の2つがポイントになります。
- ORF全体を大腸菌のコドン使用頻度に基づいたコドンを使用する
- N末端のアミノ酸は、大腸菌のコドン使用頻度に関わらず、ATリッチになるようなコドンを使用する
2.5’UTRを構成する領域とタンパク質合成量の関係について
現在、PUREfrex用に使用する5’UTRは、PUREfrexが転写酵素としてT7 RNA polymerase を使用していることから、T7ファージのgene10の5’UTRを推奨しています。(下図の control )
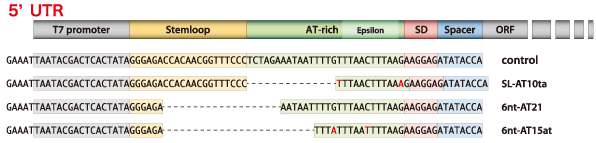
T7ファージのgene10の5’UTR配列は、T7プロモーター配列以下、主に以下の3つの領域に分かれています。
- Stemloop:stemloop構造を形成することが予想される領域
- AT-rich:AT含量が高い領域
- SD:リボソーム結合領域
今回は、鋳型DNAのアミノ酸配列だけではなく、塩基配列にも注目し、特に5’UTR部分について、PUREfrex を用いた合成に、最適な塩基配列の検討を行いました。
3.5’UTR の配列でタンパク質の合成量に影響をおよぼす領域
配列を様々に変えた5′ UTRにGFPの遺伝子配列を結合した鋳型DNAを作成し、PUREfrexで合成した後、合成されたGFPの蛍光量を比較することで、タンパク質の合成量に影響をおよぼす領域を調べていきました。
詳細につきましては、第93回 日本生化学会大会で発表したWeb動画をご覧ください。(約10分)

