Hisタグ付きのNanodisc上で膜タンパク質を合成させ、アフィニティ精製のワンステップで、反応液から膜タンパク質を精製できることを確認しました。
1. 鋳型DNAの配列最適化
1-1. プロトコル
- 使用したキット:PUREfrex®2.0(#PF201-0.25)
- 目的のタンパク質:CLDN1(Homo sapiens)
- 分子量:22,744 Da(211 a.a.)
- 反応液量:10 µL
- 合成温度:37 °C
- 合成時間:4 時間
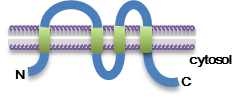
鋳型DNAは、開始コドン直後に、CLDN1の2Aspから211Valで構成されています。その内、2から6番目のアミノ酸配列を最適化しています。
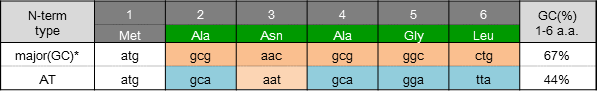
* major (GC)は、大腸菌のメジャーコドンを選択した配列になっています。
1-2. 結果
CLDN1のN末端配列をATリッチにした結果、大腸菌のメジャーコドンに合わせた配列よりも、合成量が3倍以上得られたため、ATリッチになるよう配列を最適化した鋳型DNAを用いて、次の合成と精製を行いました。
(SDS-PAGE:変性処理 37°Cで1時間、1 µL apply、CBB染色)

2. 膜タンパク質の合成
2-1. プロトコル
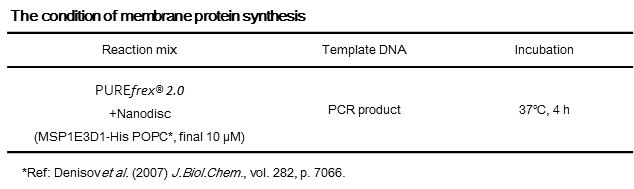
- 使用したキット:PUREfrex®2.0(#PF201-0.25)
- 使用したNanodisc:MSP1E3D1-His POPC (Cube Biotech, #26353)
- 鋳型DNA:N末の配列を最適化したCLDN1(2Ala – 211Val)
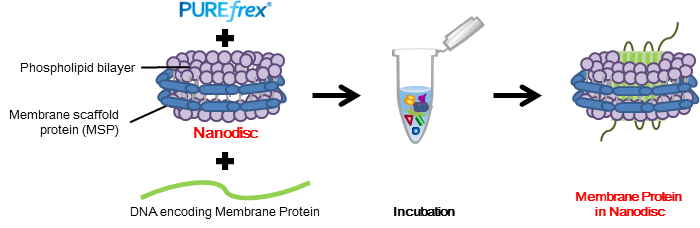
2-2. 結果
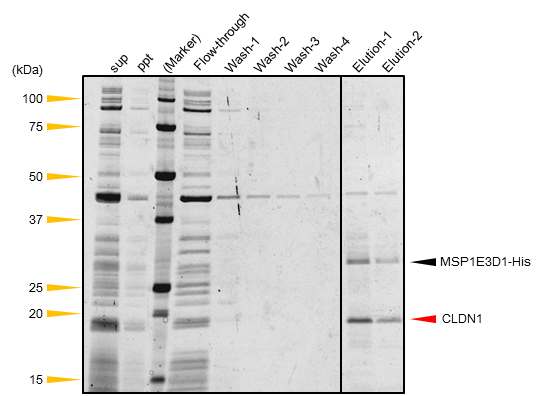
PUREfrex®2.0にNanodiscを添加し、膜上でCLDN1を合成することで、可溶化したCLDN1を得ることができました。

- 遠心条件:20,000 xg、30分、4 °C
- 変性処理:37 °Cで1時間
- SDS-PAGEアプライ量:1 µL
- ゲル染色:CBB
3. 膜タンパク質の精製
3-1. プロトコル
- 使用したキット:PUREfrex®2.0(#PF201-0.25)
- 使用したNanodisc:MSP1E3D1-His POPC (Cube Biotech, #26353)
- 鋳型DNA:N末の配列を最適化したCLDN1(2Ala – 211Val)
- Ni-磁気ビーズ:GE社製 Ni sepharose 6 fast flow
- Binding buffer(BB):50 mM Tris-HCl pH8, 500 mM NaCl, 18 mM MgCl2*, 20 mM imidazole
- Wash buffer (WB): 50 mM Tris-HCl pH8, 500 mM NaCl, 20 mM imidazole
- Elution buffer(EB):50 mM Tris-HCl pH8, 500 mM NaCl, 400 mM imidazole *)リボソームは、Buffer中のMgの濃度が低下すると、複合体が壊れてしまうため、BBにMgを添加することで、複合体を形成させた状態を維持させるよう工夫しています。
- 100 µLの反応液 (Nanodiscを同時に添加, final 10 µM) で37°C、4時間合成
- BB 800 µLを step1 の反応液へ添加
- Ni-磁気ビーズ(あらかじめBBで洗浄した50%懸濁液)200 µLを step2 に添加
- 4°Cで1時間ローテーション
- ビーズをWBで洗浄
- EBで溶出
3-2. 結果
Nanodisc上で合成したCLDN1は、アフィニティ精製のワンステップで効率的に精製することができました。
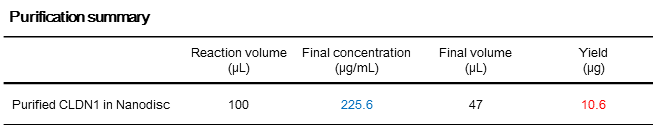
N末端の配列をATリッチにすることで、タンパク質の合成量が改善されます。他のタンパク質について調べた結果をポスター発表しておりますので、こちらもご覧ください。

