鋳型DNAの配列に関する6つの注意点
鋳型DNAの塩基配列やアミノ酸配列が原因で、合成されるタンパク質の量が低下することがあり、原因となる配列を最適化することで、タンパク質の合成量が改善されることがあります。
PUREfrex®は、リボソームやtRNAなどの翻訳に関連する因子が大腸菌由来であるため、目的タンパク質の遺伝子全体を、大腸菌での翻訳に適したコドンで最適化することをおすすめします。最適化する場合は、弊社の「鋳型DNA設計のサポート」や、様々なメーカーから提供されている最適化ツールをご利用ください。大腸菌用にコドンを最適化した後に、さらに、PUREfrex®用に修正すべき点がありますので、下記の項目についてご確認ください。
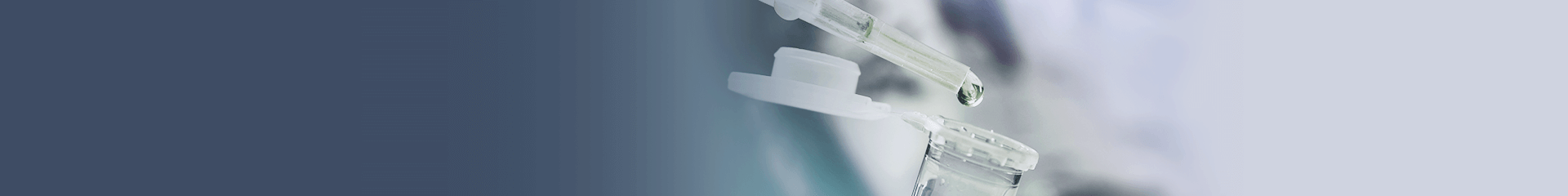
1.開始コドン直後をATリッチにする
開始コドン(ATG)直後の2番目から6番目のアミノ酸のコドンは、できるだけATが多くなるようなコドンを選択してください。この領域に限っては、大腸菌のコドンの使用頻度よりも優先した方が、タンパク質の合成量が高くなる傾向にあります。
下図に、開始コドン(ATG)直後の2番目から6番目のコドンについて、異なる組み合わせで鋳型DNAを作製し、それぞれの鋳型DNAからタンパク質の合成を行い、その合成量を比較した結果を示しています。その結果、同じタンパク質にも関わらず、開始コドン直後の2番めから6番目のコドンが、合成量に大きく影響を及ぼしていることがわかります。
2.メジャーコドンのみの偏った使用を避ける
塩基配列を大腸菌用に最適化した場合、大腸菌で使用頻度の高い1つのコドンのみが使用される場合があります。例えば、ロイシンでCTGコドンのみが使用されている場合があります。このようなDNAを用いてPUREfrex®で合成した場合、合成量が低くなる場合があります。使用されるコドンが極度に偏っている場合には、大腸菌のコドンの使用頻度に応じて適度に割り振ります。
E.coli K12株のコドン使用頻度はこちらも参照ください。
https://www.kazusa.or.jp/codon/cgi-bin/showcodon.cgi?species=83333&aa=1&style=N
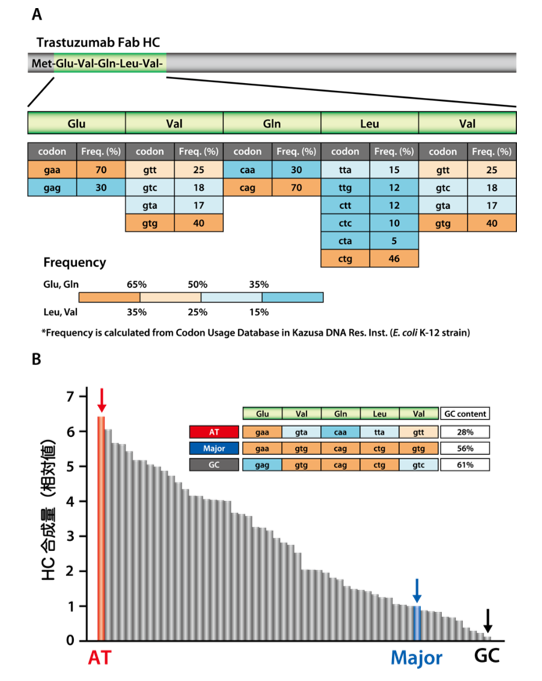
3.5′ UTR~N末端近傍の二次構造形成を避ける
SD配列付近~N末端近傍(開始から10アミノ酸程度)をコードする領域で、mRNAが強固な二次構造を形成してSD配列がマスクされるとリボソームが結合しにくくなり、合成量が低下する可能性があります。この領域で強い二次構造形成が予測される場合には、コドンを置換して塩基配列を調整してください。
二次構造予測の例として、本キットの陽性コントロールとして添付されているDHFR DNAについて、SD配列から6残基までの領域(図Aの黄色でハイライトした部分)をRNAfold server(http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi)で解析した結果を下図に示します。図Bの結果で示すように、SD配列(AAGGAG)部分が二次構造を取らないため、合成量は低下しません。
4.開始Met直後のProやGlyを避ける
開始メチオニン直後の2番目や3番目のアミノ酸がプロリンやグリシンの場合、合成量が低下する場合があります。除去可能であれば、この領域のプロリンやグリシンは避けてください。
下図に、開始メチオニン直後にグリシンがない場合と、ある場合で、合成量を比較した結果を示しています。その結果、開始メチオニンの直後にグリシンがある場合は、合成量が下がることがわかります。
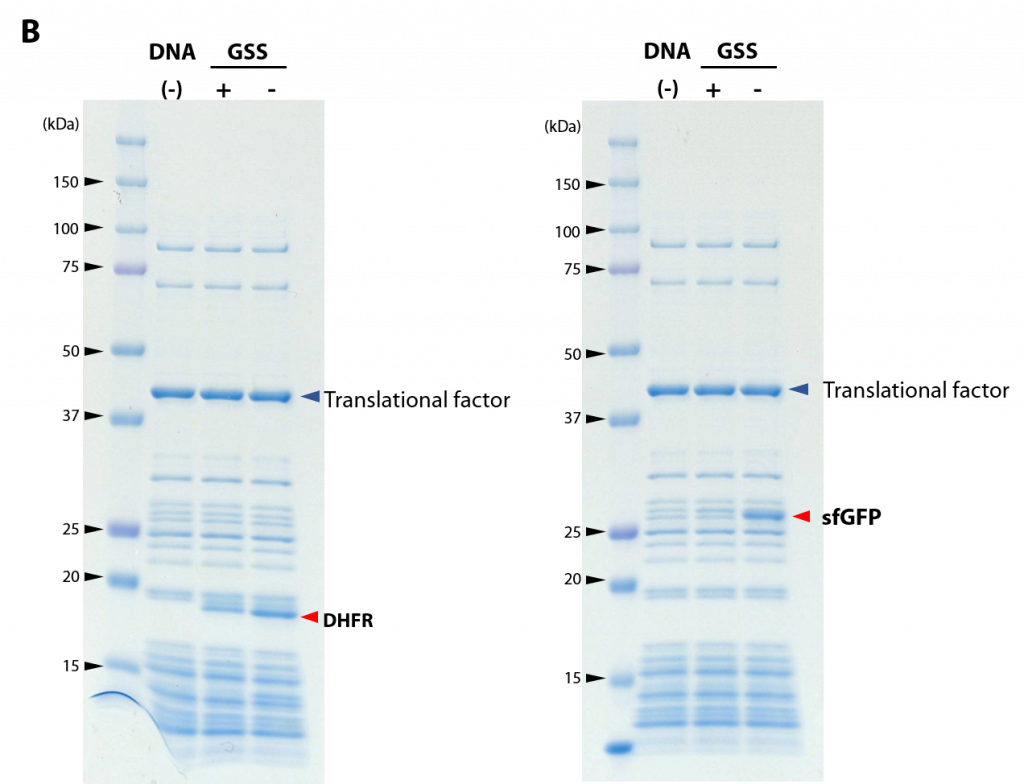
5.フレームシフトを生じさせる配列を避ける
X/XXY/YYZのようなフレームシフトを起こしやすい配列が存在する場合は、他のコドンに置き換えてください。例えば、リジンが2個連続し、塩基配列が「A/AAA/AAA」のような場合、「A/AAG/AAA」に置換するとフレームシフトが抑制されます。
<参考文献> Sharma V. et al. (2014) Nucleic Acids Res., vol.42, p.7210
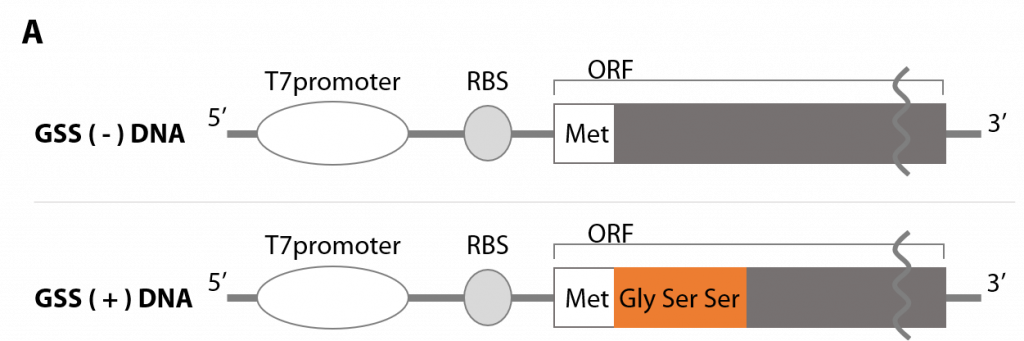
6.Proが連続する配列がある場合はEF-Pを添加する
連続したプロリンを含むタンパク質を合成すると、合成量が低いことがあります。このような連続したプロリンを含むタンパク質の翻訳において、大腸菌ではEF-Pと呼ばれる翻訳因子が関与していることが知られていますが、PUREfrex® にはEF-Pが含まれていないため、合成量が低くなる可能性があります。
PUREfrex® 2.0 にEF-Pを添加して合成した場合に、タンパク質の合成量が増大した結果について、下記のポスターをご参照ください。
https://www.genefrontier.com/files/p21_MBSJ2018.pdf